AIエンジニアやプログラマーに転職して、生命科学の未踏領域に挑戦しましょう。
DNAの研究に使用されるIT技術とPythonの具体的なコードを紹介します。
AIエンジニアとしてDNA研究の応用例も紹介するので、新しいビジネスアイデアも得られますよ。
バイオインフォマティクスとDNA研究の新展開
「遺伝子が生命の設計図ではないことを認める時」という記事では、生物学の現在の一般的な見方が過度に単純化されており、時代遅れであると述べています。
2001年にヒトゲノムが解読された時、多くの人がそれを生命の「取扱説明書」と考えましたが、実際には遺伝子だけが人間の設計図ではないそうです。
遺伝子は、食事や環境など、多くの外部要因に影響されます。また、一つの特徴は多くの遺伝子によって影響を受けることがあるということです。
科学者たちは、これまでの考え方に固執するのではなく、新しい発見に基づいてアイデアを進化させる必要があると述べています。
バイオインフォマティクス:PythonとAI技術
遺伝子だけが人間の設計図ではなく、食事や環境なども影響する、ということですね。
この記事の遺伝子研究に使用されているIT技術について推測してみましょう。
- プログラム言語:
- Python: データ分析、機械学習モデルの構築、バイオインフォマティクスのスクリプティングに広く使用されます。
- R: 統計分析やデータの可視化に特化した言語で、生物学的データの解析によく使われます。
- Java: 大規模なバイオインフォマティクスアプリケーションの開発に使われることがあります。
- AI技術:
- 機械学習: 遺伝子発現データやタンパク質の相互作用のパターンを解析するために使用されます。
- ディープラーニング: 複雑な生物学的システムのモデリングや、画像ベースのデータ(例: 細胞の画像)の解析に利用されます。
- クラウド技術:
- AWS (Amazon Web Services): 大量の遺伝子配列データの保存や計算に使用されます。
- Google Cloud Platform: バイオインフォマティクスのワークフローをサポートするサービスを提供します。
- Microsoft Azure: 生命科学の研究で使用される、さまざまな計算資源やAIツールを提供します。
- データ分析ツール:
- Bioconductor: Rを用いた高度なゲノミクスデータ解析に使用されるツール群です。
- Galaxy: バイオインフォマティクス解析を行うためのウェブベースのプラットフォームで、コーディング知識がない研究者にも使いやすいです。
各技術やツールは、遺伝子を理解するために不可欠です。
Pythonで実践!DNA配列解析のサンプルコード
Pythonを使ってDNAの研究に応用できる機械学習のサンプルコードを紹介しましょう。
ここでは、DNA配列の特定パターン(例えば、特定の遺伝子が発現するかどうか)を識別する分類器を構築するというシナリオを想定します。
サンプルデータとして、DNA配列を模擬したデータを使用します。
import numpy as np
import pandas as pd
from sklearn.model_selection import train_test_split
from sklearn.ensemble import RandomForestClassifier
from sklearn.metrics import accuracy_score, confusion_matrix, classification_report
import matplotlib.pyplot as plt
import seaborn as sns
# Sample DNA sequences and their labels (0 = non-expressive, 1 = expressive)
data = {
'Sequence': ['ATCG', 'GCTA', 'CGAT', 'TAGC', 'CCGG', 'GGCC', 'AATT', 'TTAA', 'CAGT', 'TCAG'],
'Label': [0, 1, 0, 1, 0, 1, 0, 1, 0, 1]
}
df = pd.DataFrame(data)
# Convert DNA sequences to numerical data (simple encoding: A=0, T=1, C=2, G=3)
def encode_sequence(seq):
mapping = {'A': 0, 'T': 1, 'C': 2, 'G': 3}
return [mapping[char] for char in seq]
df['Encoded'] = df['Sequence'].apply(encode_sequence)
# Split the dataset into training and testing sets
X_train, X_test, y_train, y_test = train_test_split(df['Encoded'].tolist(), df['Label'], test_size=0.3, random_state=42)
# Random Forest Classifier
clf = RandomForestClassifier(n_estimators=100, random_state=42)
clf.fit(X_train, y_train)
# Predictions
y_pred = clf.predict(X_test)
# Evaluation
accuracy = accuracy_score(y_test, y_pred)
print(f'Accuracy: {accuracy}')
conf_matrix = confusion_matrix(y_test, y_pred)
print('Confusion Matrix:\n', conf_matrix)
# Plotting Confusion Matrix
plt.figure(figsize=(5,4))
sns.heatmap(conf_matrix, annot=True, fmt='g')
plt.xlabel('Predicted labels')
plt.ylabel('True labels')
plt.title('Confusion Matrix')
plt.show()
# Classification Report
report = classification_report(y_test, y_pred)
print('Classification Report:\n', report)Accuracy: 0.3333333333333333
Confusion Matrix:
[[1 0]
[2 0]]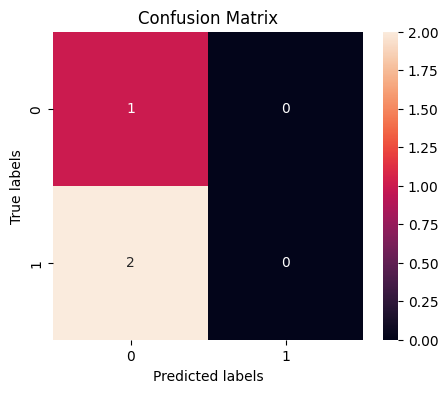
Classification Report:
precision recall f1-score support
0 0.33 1.00 0.50 1
1 0.00 0.00 0.00 2
accuracy 0.33 3
macro avg 0.17 0.50 0.25 3
weighted avg 0.11 0.33 0.17 3このコードは、次の機能を実装しています。
- データの準備: DNA配列のサンプルとそれに対応するラベル(発現するかどうか)を作成します。この例では、4文字の配列を使っていますが、実際の応用ではもっと長い配列が一般的です。
- 配列のエンコーディング: DNA配列を数値データに変換するため、シンプルなエンコーディング手法を採用しています(A=0, T=1, C=2, G=3)。実際の研究では、より複雑なエンコーディングが必要になる場合があります。
- 訓練データとテストデータの分割: 機械学習モデルを訓練するためにデータセットを訓練セットとテストセットに分割します。
- ランダムフォレスト分類器の使用: 分類問題を解決するために、ランダムフォレストアルゴリズムを使用します。ランダムフォレストは、多数の決定木を組み合わせることで、分類の精度を向上させるアンサンブル学習の一種です。
- モデルの評価: テストデータセットに対する予測を行い、精度(Accuracy)、混同行列(Confusion Matrix)、分類レポートを用いてモデルの性能を評価します。
- 混同行列の可視化: 混同行列をヒートマップとしてプロットし、モデルの予測がどの程度正確であるかを視覚的に確認します。
上記のPythonコードはあくまでサンプルであり、実際のDNA配列データを扱う場合には、データの前処理、モデルの選択、パラメータのチューニングなど、より詳細なステップが必要です。
バイオインフォマティクスの応用
「PythonでDNAの研究」について、同業種や他業種へ応用できるアイデアを考えてみます。
同業種への応用アイデア
- バイオインフォマティクスのスキル向上:
- DNA配列解析の技術を習得し、バイオインフォマティクス分野でのプロジェクトに貢献する。
- カスタム解析ツールの開発:
- 研究者のニーズに応えるための特定のDNA解析を行うカスタムソフトウェアやツールを開発する。
- 機械学習の適用:
- DNAデータを用いて、遺伝子発現のパターンや疾患のリスク予測などを行う機械学習モデルを構築する。
他業種への応用アイデア
- データ解析の技術転用:
- DNA解析で用いるデータ処理技術を、金融やマーケティングなどのデータ集約型業界でのビッグデータ解析に応用する。
- アルゴリズムの応用:
- 生物学的データを解析するために開発されたアルゴリズムを、画像処理や自然言語処理など他の領域の問題解決に適用する。
- クラウドコンピューティングの活用:
- DNA解析で使用するクラウドベースの計算資源や分散処理の技術を、他の業界での大規模データ処理やリアルタイム分析に応用する。
DNAの研究で用いられるプログラミング言語や技術は、さまざまな業界や研究分野で応用できる可能性がありますね。
まとめ:AIエンジニアとDNA研究が拓く新しい世界
「DNAは人間の設計図の全てではない」という記事から、研究に使われるIT技術、Pythonコード、応用アイデアなどを紹介しました。
DNAの研究は、私たちの生活や社会に大きな影響を与える可能性があります。
あなたもAIエンジニアやプログラマーに転職して、新しい発見やイノベーションを生み出してみませんか?

▼AIを使った副業・起業アイデアを紹介♪




